SuperStar
タンパク質-リガンド結合部位の相互作用マップ生成プログラム
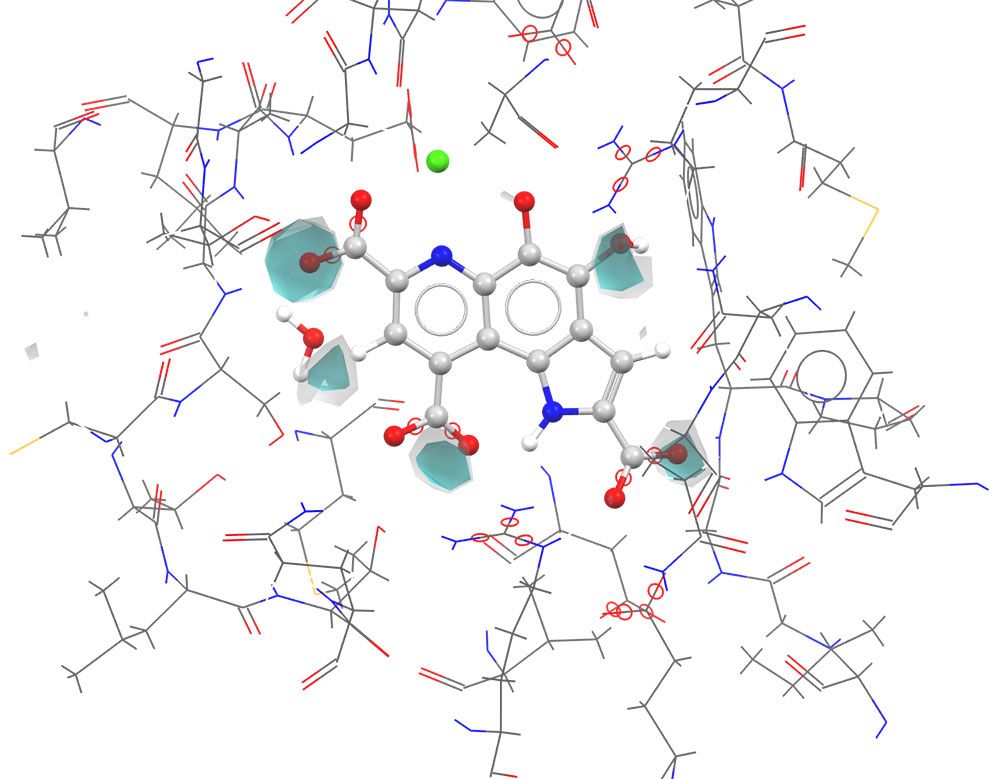
SuperStarは、ケンブリッジ結晶構造データベース(CSD)とProtein Data Bank (PDB)の結晶構造情報に基づき、タンパク質とリガンドの相互作用を予測し可視化するプログラムです。タンパク質-リガンド結合部位に対する理解が深まり、化合物デザインの精度が向上します。
CSD-Discovery および CSD-Enterprise に付属しています。
SuperStarには、どのような特徴がありますか?
実験データに基づく知識ベースの予測
SuperStarは、CCDCの分子間相互作用ライブラリであるIsoStarからデータを取得しています。
IsoStarには、CSDとPDBの相互作用情報が含まれており、knowledgeベースの相互作用マップが作成されます。
短時間でのマップ作製
IsoStarからデータを取得を取得しているため、エネルギーベースの計算よりも短時間で相互作用マップを生成することができます。
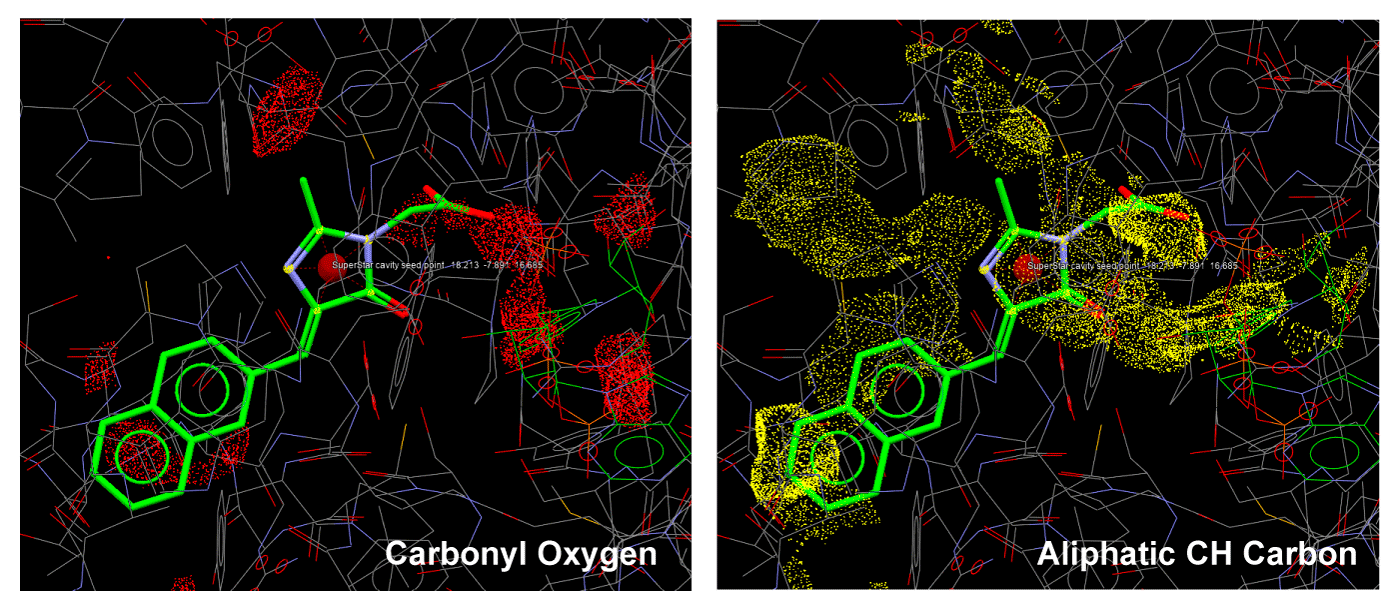
ホットスポットの特定
タンパク質とプローブ(官能基)間の相互作用の確率は、結晶構造において相互作用が観察される頻度に基づいて推定されます。
これらはプローブごとに密度マップとして可視化され、タンパク質表面のホットスポットの特定が可能になります。
ワークフローのカスタマイズ
CSD Python APIを利用して、現在のワークフローに追加することができます。
オックスフォード大のSmilovaとCCDCが報告したアンサンブルホットスポットマップのコードや “Hotspots API” のインストールに必要なデータはCCDCのGitHub repositoryでオープンソースとして公開されています。
CCDCのサイトは、こちらから。
SuperStarは、どのような場面で用いられますか?
SuperStarは、タンパク質-リガンド結合部位の相互作用マップを生成し、ホットスポットを可視化することにより、以下を可能にします。
